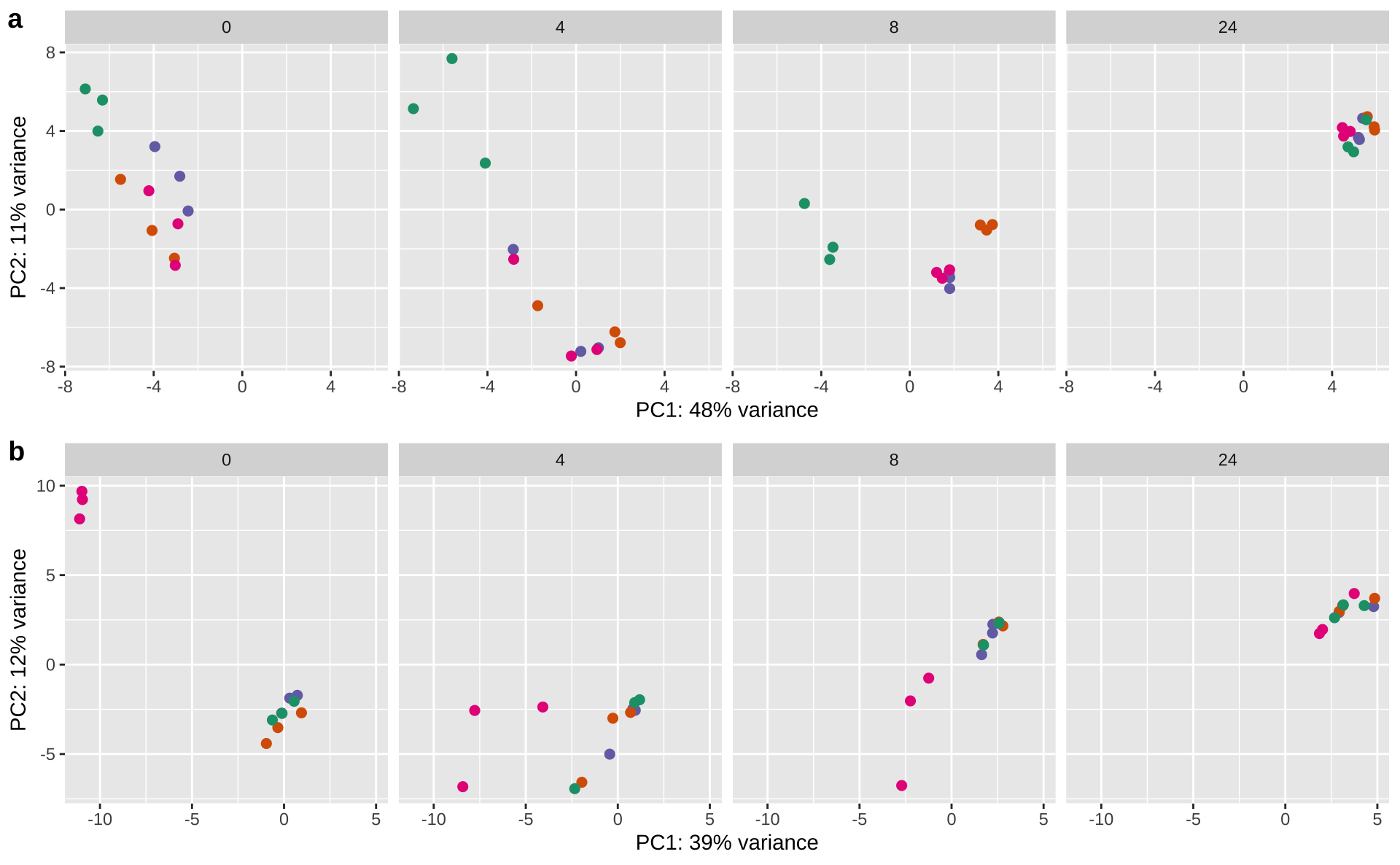
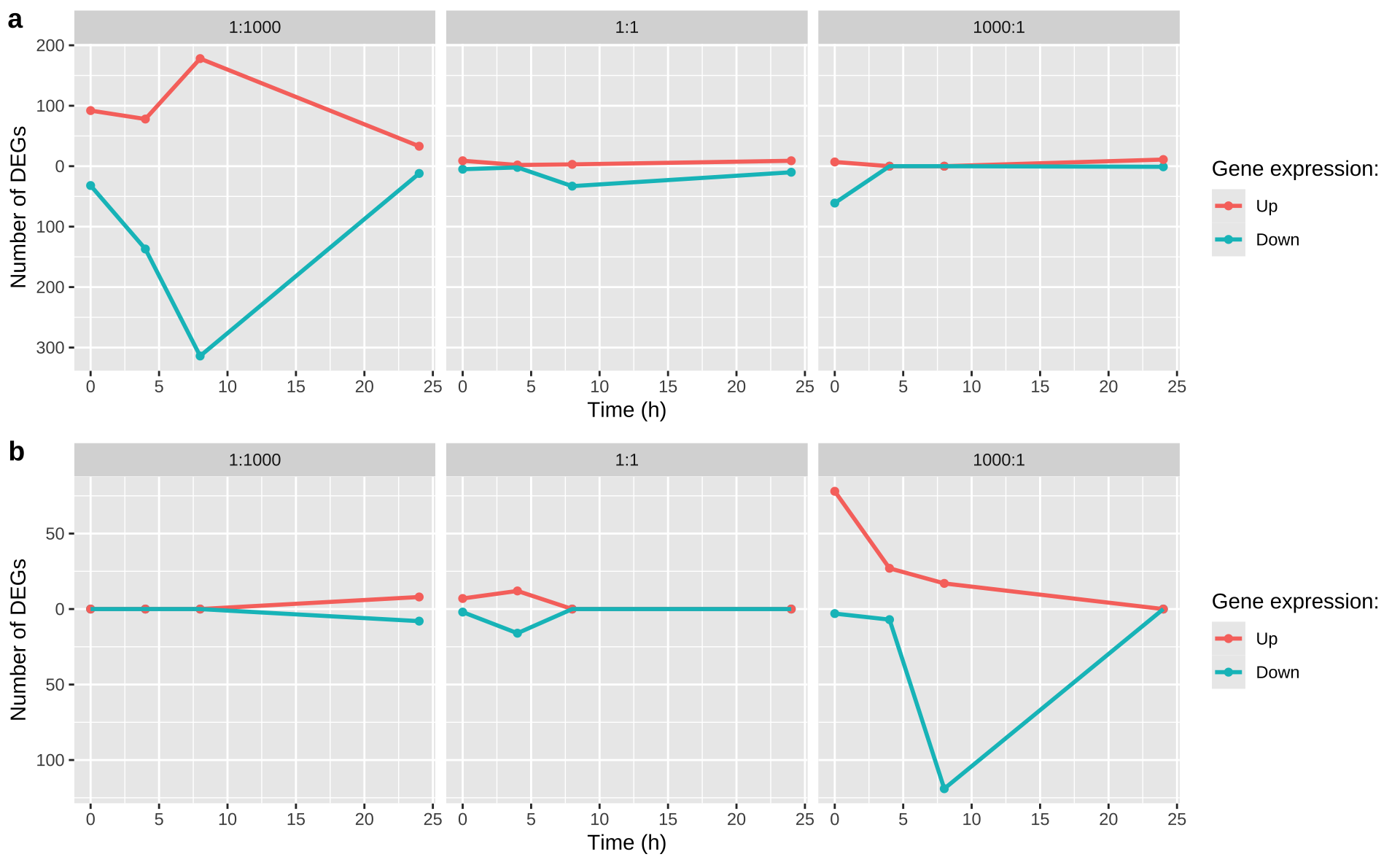
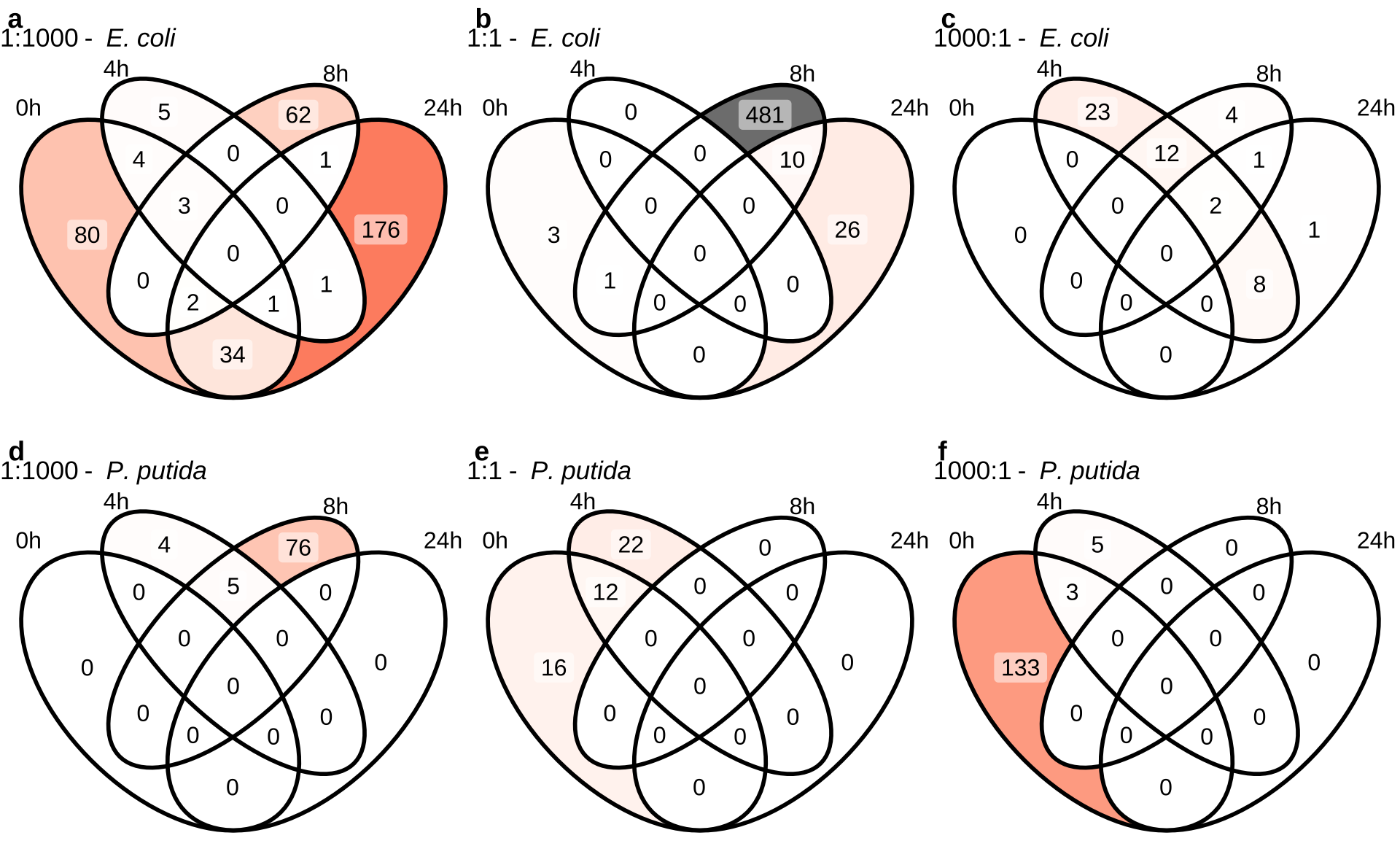
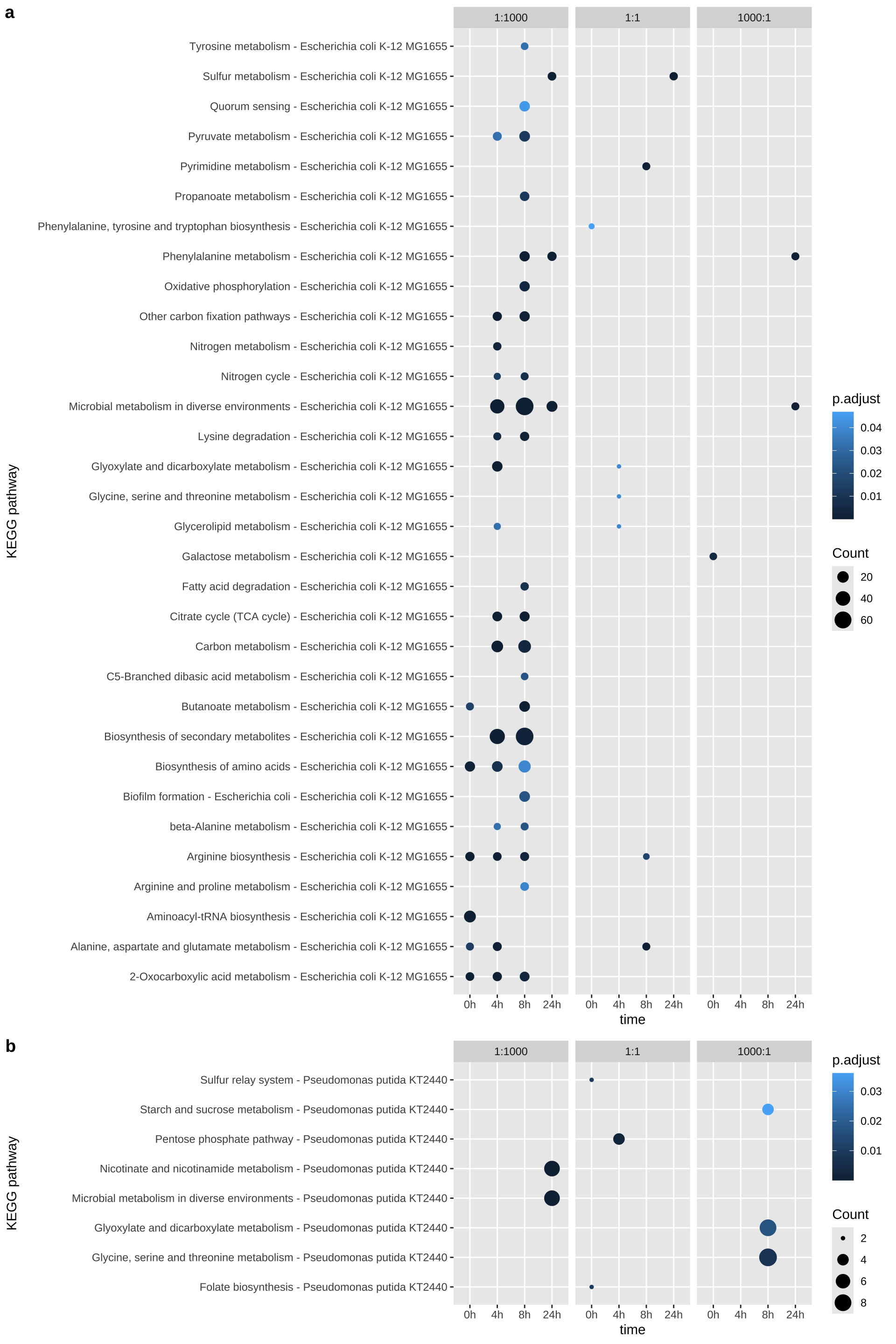
# 定义 E. coli 的比较组
comparisons.EC <- list(
c("less_0h", "all_0h"), c("equal_0h", "all_0h"), c("more_0h", "all_0h"),
c("less_4h", "all_4h"), c("equal_4h", "all_4h"), c("more_4h", "all_4h"),
c("less_8h", "all_8h"), c("equal_8h", "all_8h"), c("more_8h", "all_8h"),
c("less_24h", "all_24h"), c("equal_24h", "all_24h"), c("more_24h", "all_24h")
)
# 定义 P. putida 的比较组
comparisons.PP <- list(
c("less_0h", "none_0h"), c("equal_0h", "none_0h"), c("more_0h", "none_0h"),
c("less_4h", "none_4h"), c("equal_4h", "none_4h"), c("more_4h", "none_4h"),
c("less_8h", "none_8h"), c("equal_8h", "none_8h"), c("more_8h", "none_8h"),
c("less_24h", "none_24h"), c("equal_24h", "none_24h"), c("more_24h", "none_24h")
)
# 提取差异表达基因结果
DEG_results.EC <- myDEG_Results(dds = dds.EC, comparison = comparisons.EC)
DEG_results.PP <- myDEG_Results(dds = dds.PP, comparison = comparisons.PP)